



我院李大力、刘明耀团队合作对腺嘌呤脱氨酶TadA-8e重新设计改造,创新性地构建了一系列“精准且安全”的新型胞嘧啶碱基编辑器,编辑窗口更加精准,脱靶率极低。

李大力教授(右三)、陈亮(右二)、朱碧云(右一)、汝高盟(左二)
构建“精准且安全”的新型胞嘧啶碱基编辑器
目前已知的人类遗传疾病约60%是由碱基突变引起的[1, 2],因此开发高效精准且安全的单碱基编辑器对基础研究和人类遗传疾病的治疗具有极为重要的意义。
哈佛大学David R. Liu团队分别通过将Cas9缺口酶(nickase)与APOBEC家族胞嘧啶脱氨酶以及通过分子进化得到的腺嘌呤脱氨酶TadA融合,开发出可实现C到T碱基转换的胞嘧啶碱基编辑器(Cytosine base editor, CBE)以及A到G碱基转换的腺嘌呤碱基编辑器(Adenine base editor, ABE)[3, 4]。
这两类碱基编辑器能不依赖DNA双链断裂和同源重组修复机制,在分裂及非分裂细胞基因组DNA上实现高效的碱基转换。胞嘧啶脱氨酶催化C转变为U的过程通常伴随着尿嘧啶的切除,形成的无嘌呤无嘧啶位点最终导致潜在的C到G/A副产物的形成,因此在CBE的基础上,通过对APOBEC1的改造等策略开发了主要实现C到G碱基颠换的CGBEs[5-9]。
然而现有的CBEs/CGBEs均依赖于天然的胞嘧啶脱氨酶AID/APOBEC家族成员,由于胞嘧啶脱氨后会激活碱基切除修复通路,不可避免的会产生较高的indels副产物。更为重要的是现有的CBE会产生显著的不依赖于Cas9的DNA和RNA随机脱靶,而且较宽的编辑窗口引起难以避免的旁观者效应,在安全性等方面存在隐患[10, 11]。
华东师范大学生命科学学院李大力、刘明耀团队以及北京大学生命科学学院伊成器课题组合作通过对腺嘌呤脱氨酶TadA-8e重新设计改造,创新性地构建了一系列不依赖天然胞嘧啶脱氨酶家族的“精准且安全”的新型胞嘧啶碱基编辑器—TadA-derived CGBE/CBEs (Td-CGBE/Td-CBEs),其中Td-CGBE能高效精确地诱导C到G的转换,而进一步的分子进化获得了与BE4max活性相当但编辑窗口更窄的Td-CBEs系列。
更为重要的是,消除了固有的腺嘌呤脱氨酶活性的新型CGBEs/CBEs,展现出非常低的indels事件。通过项目组前期开发最高灵敏度CBE脱靶检测技术Detect-seq,证明Td-CBEs相较于BE4max展现接近背景水平的更低的脱靶效应。
“Td-CGBE/Td-CBEs”临床应用前景广阔
首先,该研究证明了ABE尤其是高活性ABE8e可介导高效的胞嘧啶编辑,猜测TadA-8e脱氨酶对腺嘌呤和胞嘧啶有一个共同的催化口袋,活性位点及其周围的关键氨基酸改变可能会影响催化底物的选择。
基于结构导向的理性设计和筛选,发现在TadA-8e脱氨酶的活性口袋引入关键突变N46L,消除其固有的腺嘌呤脱氨酶活性而展现高效的胞嘧啶编辑活性,随后在27个靶点(包括4个富含多A的靶点)的测试进一步证明了该突变可完全消除腺嘌呤编辑活性,诱导高效率高纯度的C到G的转换(效率高达72.8%)。
令人惊喜的是,其相对于传统的CGBE编辑器,该突变体具有更低的indels,且编辑窗口显著缩窄到1-2个碱基 (C5-C6)。由此开发了首款不依赖于天然AID/APOBEC家族酶的CGBE,命名为TadA-derived CGBE(Td-CGBE)。(图1)
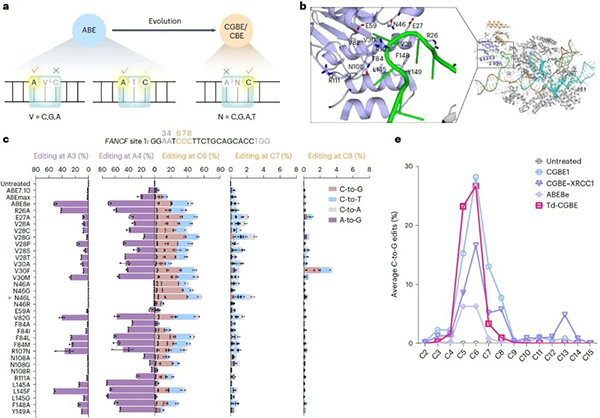
图1:TadA-8e蛋白结构及Td-CGBE特性评价
Td-CGBE高精确度的特性为进一步开发精准的C到T的碱基编辑器奠定基础,项目组通过对TadA-8e结构中可能与底物相互作用的关键区段重新改造、linker序列以及UGI串联等多轮优化获得了一系列Td-CBEs,发现其依然表现为完全消除腺嘌呤编辑的特性,其中TadA-8e引入关键突变E27R 获得的Td-CBEmax,在13个靶点测试后发现,其保留高效的C到T编辑活性,产生与BE4max相似的编辑活性(57.7%-94.9%),但编辑窗口显著缩小到sgRNA的5-7位,而且产生显著降低的indels频率。
为了解决传统CBE的精准性问题,该研究发现在Td-CBE的基础上去除linker序列并在TadA-8e-N46L的基础上引入P29A(eTd-CBEa)或A48M(eTd-CBEm)可保留较高的C到T编辑活性,并且显著缩小编辑窗口到sgRNA的第5或第6位,与基于APOBEC1来源的窄窗口BE4max-YEE相比,eTd-CBEs变体的精确度最高提升了131.4倍,且仅诱导背景水平的indels事件(图2)。
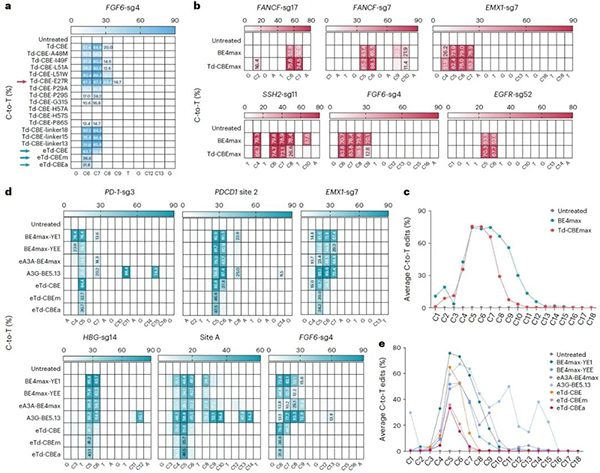
图2:Td-CBEs的优化及特性评价
通过Cas9依赖性的脱靶位点、Cas9非依赖性脱靶的R-loop检测和项目组前期开发的最灵敏的Detect-seq以及RNA脱靶的转录组分析,令人兴奋的是,Td-CGBE/Td-CBEs变体几乎不引起随机的DNA和RNA脱靶编辑(图3),展示出了极高的应用安全性。
此外,Td-CGBE在小鼠胚胎中展示出极高的体内编辑活性和精度。在所有的F0代小鼠中,Td-CGBE可以高效编辑靶位点的C5位碱基(20/21只),其中67%的小鼠产生C到G的编辑,最高效率达到84.9%,相对于APOBEC1来源的CGBE有更高的效率且诱导极低的indels。
为进一步评估Td-CGBE/Td-CBEs变体基因治疗的潜能和其精准的特性,研究者评估了其在背景序列含有多个同源多聚胞嘧啶位点的致病单核苷酸变异(SNVs)的编辑情况,这些位点中需要纠正的胞苷处于sgRNA第五位且处于多个连续胞苷中。
发现Td-CGBE/Td-CBEs能精准编辑sgRNA第五位的胞苷并产生所需的C-to-G/T转换,相对于传统CGBE/CBEs展现出更精准、高效地构建或纠正致病 SNVs位点的能力(图4)。
最后,为了无偏见地分析eTd-CBEs的精准编辑特性,该研究采用含有8954条sgRNA与靶点配对的文库进行编辑窗口的分析,发现eTd-CBEm非常精准且无序列偏好地主要编辑C5位碱基,证明其广泛地精准编辑特性(图4)。由于其无编辑序列偏好,因此将来还可以与识别不同PAM的Cas蛋白变体融合,进一步扩大靶向范围。
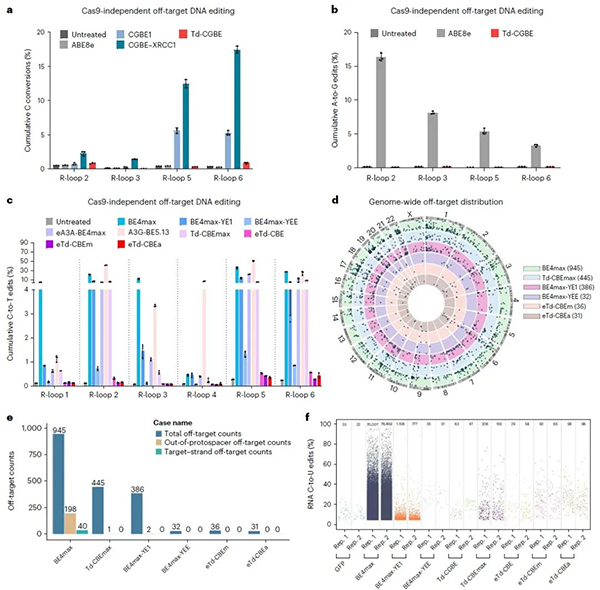
图3:Cas9非依赖DNA脱靶、detect-seq和RNA脱靶评价
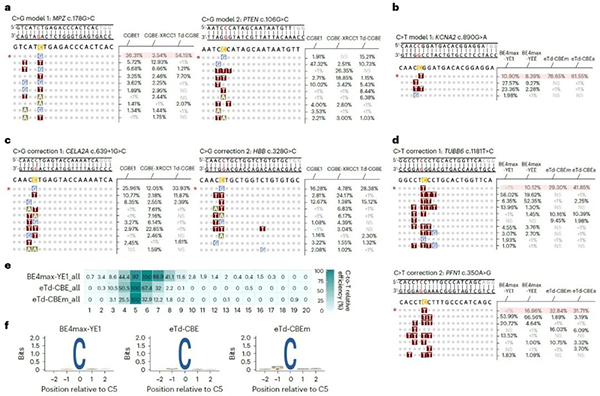
图4:Td-BEs变体纠正人类致病性的SNVs及靶向文库分析无偏见的评价eTd-CBEs编辑特性
总的来说,该研究创造性地通过蛋白改造,将以腺嘌呤为底物的脱氨酶TadA-8e改造成为仅以胞嘧啶为底物的非天然的胞嘧啶脱氨酶,并构建了第一个不依赖AID/APOBEC 脱氨酶家族的新型CGBE/CBE系列碱基编辑器-Td-CGBE/Td-CBEs,表现出与BE4max系列编辑器相当的编辑效率,而编辑窗口更加精准,脱靶率极低(接近背景水平)。
该研究一方面为深入理解TadA-8e的结构和工作原理从而开发新的碱基编辑技术提供了新思路,更为重要的是有望极大提高将来临床应用的安全性。碱基编辑器的优化升级及安全性的完善为人类遗传疾病的基因治疗带来希望。

研究成果以题为Re-engineering the adenine deaminase TadA-8e for efficient and specific CRISPR-based cytosine base editing的研究论文在Nature Biotechnology杂志上发表。
华东师范大学生命科学学院博士研究生陈亮、朱碧云,硕士研究生汝高盟,以及北京大学生命科学学院博士后孟浩巍,博士研究生闫永昌为该论文的共同第一作者,华东师范大学为第一单位,华东师范大学生命科学学院李大力教授,刘明耀教授和北京大学生命科学学院伊成器教授为本文通讯作者。
该研究受到了科技部重点研发计划、国家自然科学基金以及上海市教委前沿科学基地和重大项目等支持。
参考文献:
[1] Landrum MJ, Lee JM, Riley GR, et al. Clinvar: Public archive of relationships among sequence variation and human phenotype. [J]. Nucleic acids research, 42, 2014, 42: D980-D985
[2] Rees HA, Liu DR. Base editing: Precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet,2018, 19: 770-788
[3] Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533: 420-424
[4] Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature, 2017, 551: 464-471
[5] Koblan LW, Arbab M, Shen MW, et al. Efficient C*G-to-G*C base editors developed using crispri screens, target-library analysis, and machine learning. Nat Biotechnol, 2021, 39: 1414-1425
[6] Kurt IC, Zhou R, Iyer S, et al. Crispr c-to-g base editors for inducing targeted DNA transversions in human cells. Nat Biotechnol, 2021, 39: 41-46
[7] Yuan T, Yan N, Fei T, et al. Optimization of c-to-g base editors with sequence context preference predictable by machine learning methods. Nat Commun, 2021, 12: 4902
[8] Chen L, Park JE, Paa P, et al. Programmable c:G to g:C genome editing with crispr-cas9-directed base excision repair proteins. Nat Commun, 2021, 12: 1384
[9] Zhao D, Li J, Li S, et al.Glycosylase base editors enable c-to-a and c-to-g base changes. Nat Biotechnol, 2021, 39: 35-40
[10] Jin SA-O, Zong YA-O, Gao QA-O, et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science, 2019, 364: 292-295
[11] Zuo E, Sun Y, Wei W, et al.Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science, 2019, 364: 289-292